人类正常细胞的综合甲基化图谱

▲ 图片 Credit: shuoshu/DigitalVision Vectors/Getty
了解细胞的 DNA 甲基化状态对于理解细胞的基因表达模式和细胞表型之间的关联具有至关重要的意义。然而,大多数已有的人类 DNA 甲基化数据集,都仅覆盖了人类基因组中的 3000 万个甲基化位点中的一小部分,并且仅限于体外培养的细胞或含有未准确定义的细胞混合物的大块组织。
近日,Loyfer 等人在《Nature》杂志上发表了一份公开的人类甲基组的综合图谱,以及用于分析混合样本的细胞类型特异性标记的计算工具,为人们进一步理解基因组甲基化的模式、发现其发育与病理相关的改变,提供了丰富的数据和工具。

该图谱是使用来自健康成年人的纯化细胞群的全基因组亚硫酸氢盐测序 (WGBS) 构建而成,代表了来自 137个人类个体的 205个样本中的 77种原代细胞类型。作者还开发了分析 WGBS 数据的软件工具,作者使用该软件将基因组分割为 2,783,421个“甲基化模块” (涵盖了至少 3个相邻的 CpG位点),这些“甲基化模块”在细胞类型之间存在着差异。值得注意的是,对甲基化模块的关注还反映了 DNA甲基化的区域性质,而这是以前基于芯片阵列的数据集所无法实现的,因为这些数据集仅可描绘单个 CpG位点。
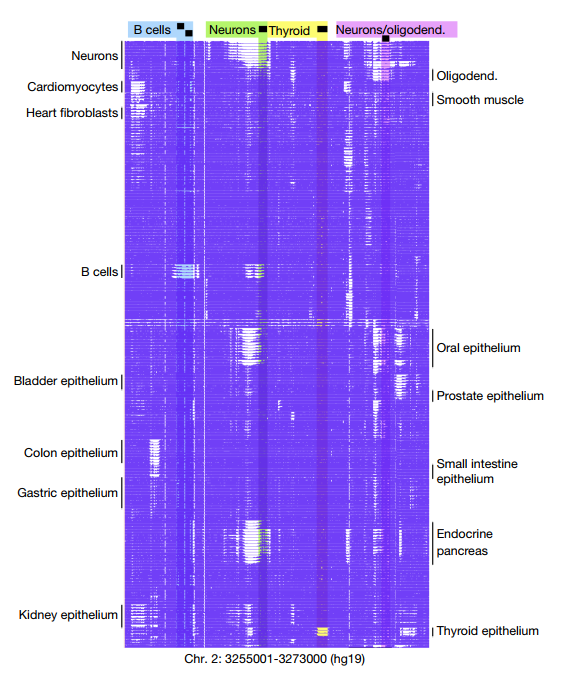
▲ 图1 | 成人人体的甲基化图谱。
甲基化模块的模式在生物重复样本之间具有高度的相似性,这种相似性支持了甲基化状态主要由细胞谱系决定,而非遗传或环境因素决定的遗传法则。此外,甲基化模式可以用来确认细胞类型之间的发育关系。作者使用无监督凝聚算法对 205个样本进行了聚类分析,根据 20997个样本间变异最大的模块的甲基化状态作者得到了扇形聚类图。在其聚类关系中,不仅可将相同细胞类型的生物样本聚集在一起,而且还可以概括已知的谱系关系。例如,胰岛细胞与胰管细胞和腺泡细胞聚集在一起,然后与肝细胞聚集在一起,因为它们都是内胚层起源的。
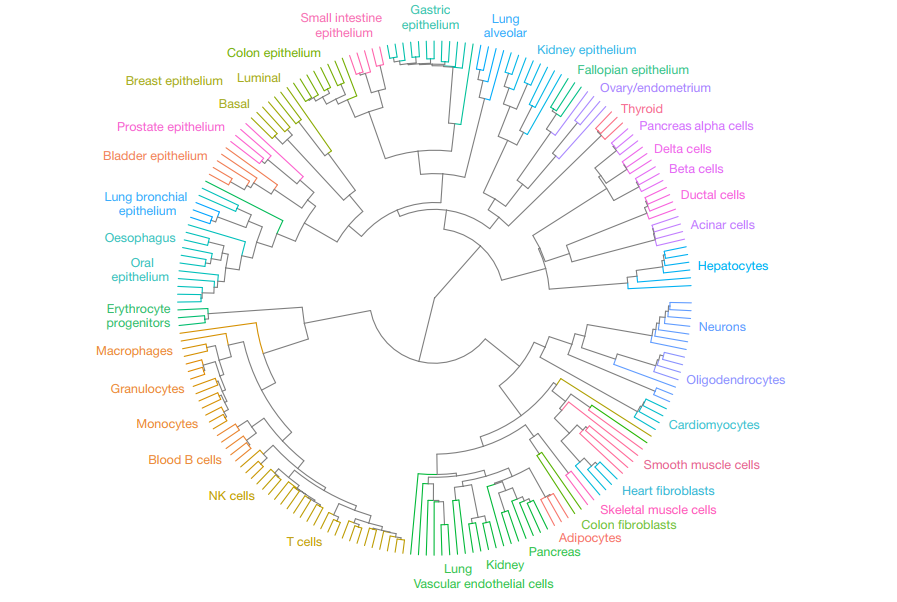
▲ 图2 | 无监督凝聚聚类反映了健康细胞类型的人类发育谱系。
为了识别细胞类型特异性标记,作者将样本分为 39组相同的细胞类型,并着重分析了其中一组与所有其他各组之间的甲基化差异 (主要是未甲基化) 的模块。每种细胞类型的前 25个差异未甲基化区块共同组成了 953个细胞类型特异性甲基化标记的图谱,这在分析混合组织样本和无细胞 DNA (cfDNA) 方面具有巨大的潜力。接着,作者利用这些细胞类型特异性的标记开发了一种 DNA 甲基化测序数据的反卷积算法,并证实了使用 DNA 序列混合物推断细胞类型组成的方法的准确性。与之前使用的有限的甲基组数据的研究相一致的是,来自健康献血者血液的 cfDNA 主要来自白细胞,少量来自血管内皮细胞和肝细胞。令人惊讶的是,新的图谱也显示了巨核细胞和红细胞祖细胞对血液 cfDNA 的显著贡献。对 52例 COVID-19 住院患者的 WGBS 数据使用相同的算法,作者发现血管内皮细胞对与疾病严重程度相关的 cfDNA 有显著贡献。这种对 COVID-19 和其他疾病中 cfDNA 的分析可以为许多病理中的组织特异性损伤提供新的见解。
为了进一步表征差异甲基化区域,Loyfer 等人通过基因集分析表明,这些区域附近的基因主要反映细胞类型特异性功能。他们进一步的研究表明,细胞类型特异性的非甲基化区域具有高水平的 DNA 可及性,并且富含组蛋白标记,提示着启动子 (H3K27ac) 和增强子 (H3K4me1) 的活性。为了与差异未甲基化区域代表基因增强子的假设保持一致,作者使用了一种计算算法来识别在细胞类型特异性标记未甲基化的地方表达增加的附近基因。作者经过分析,确定了许多细胞类型的标志基因,如胰岛标记的胰岛素和胰高血糖素基因。对 39种细胞类型中每种细胞类型的未甲基化基因组区域进行映射,用于生成细胞类型特异性假定增强子区域的目录,以供进一步分析。
而大多数甲基化差异区域在感兴趣的细胞类型中是未甲基化的,其中约 3%在一种细胞类型中是甲基化的,而在其他地方是未甲基化的。这些高甲基化区域富集了染色质环化因子 CTCF 的目标序列,这表明它们可能参与了细胞类型特异性的 3D基因组组织。将 DNA 甲基化模式与已发表的 CTCF 占据结肠和小肠中特定甲基化位点的数据进行比较,作者表明 DNA 甲基化阻止了 CTCF 在该位点结合。
因此,本研究中既提供了全面的人类甲基化组图谱,也提供了基因调控和增强子活性的机制的新见解。同时,该研究还为疾病状态下的 cfDNA 的分析,建立了新的应用方向,具有巨大的临床潜力和价值。
原文链接:https://doi.org/10.1038/s41576-023-00576-y
来源:BioMedAdv 2023-02-25